geneXplain 平台
geneXplain 平台
geneXplain 平台是一个在线工具箱和工作流管理系统,是提供生物信息学、系统生物学和化学信息学分析工具的综合性平台,适用于生命科学领域的转译研究,主要应用于个性化医学和药物基因组学。geneXplain平台整合了一系列独立模块,各个模块(即Bricks)统一在标准化界面下,外观一致,可以灵活地组合到综合工作流中。 这些模块组成的系统为转录组学和蛋白质组学提供从数据到新药物的完整分析流程。通过简单的拖放系统即可直观地管理工作流。 使用此系统,您可以编辑预定义的工作流或者从头编写自定义工作流。
geneXplain平台模块可以轻松地作为脚本或插件添加,并可与预先设定的分析结合使用。
GeneXplain GmbH为学术和商业合作伙伴提供专业服务。我们提供了许多最新的模块,一部分模块免费供用户使用,而另一部分的则需要用户支付许可证费用,用于确保平台维护和动态更新以适应该技术领域快速发展的需要。
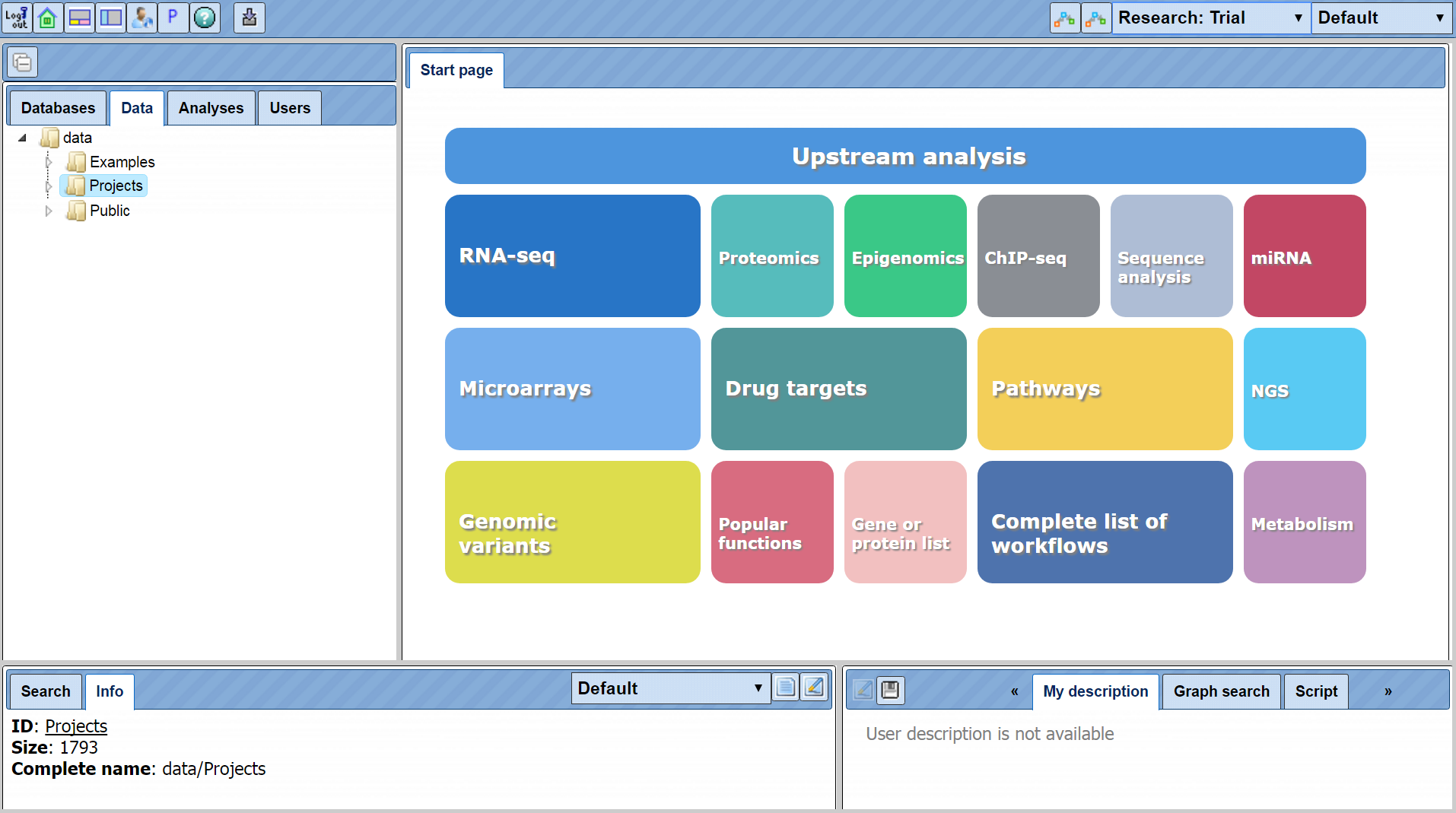
可轻松访问起始页面和多个应用程序区域。 点击图片查看全图。
关键特征
集成数据库和分析工具
该平台提供了多个数据库和分析工具(含公共领域和商业领域)的集成视图。 可以高度灵活地将它们组合在一起,用来设计客制化分析。
现成的工作流程,轻松入门
平台中提供了过百个已经验证的工作流,便于用户快速轻松地访问平台及其复杂的分析功能。 输入表格简单易用。 可以轻松地客制化工作流。 有经验的用户可以创建他们自己的工作流。
完全集成的上游分析
该平台提供了一个完全集成的上游分析,与最新的调控基因组区域分析和复杂的通路分析结合。
基于知识的数据分析
该平台使用许多著名的高质量数据库进行数据分析。 TRANSFAC®和TRANSPATH®是专家人工提取总结的数据库。 GeneWays是通过基于NLP的文本挖掘方法生成的,为手动整理的数据提供了有用的补充。 Reactome和HumanCyc等著名的公共领域数据库也被集成并应用于平台中。
JavaScript和R脚本
可以将JavaScript和R中的特定于用户的脚本直接添加到平台中,并可立即执行。 它们可以与预先设定的分析结合,成为工作流的一部分。
NGS数据分析
该平台支持NGS数据分析。 如: 包含体内转录因子结合位点或甲基化结果的ChIP-seq数据集可借助现成的工作流进行分析。平台集成了Galaxy工具,支持RNA序列数据分析,及其他多种功能。
内部模拟引擎
该平台包含一个执行微分方程系统和可视化结果的模拟引擎。 支持参数优化,参数拟合(基于表达式数据)和层次建模。
项目工作分组,包括聊天功能
与项目的其他成员共享您的数据和结果。 分析数据集时,便于用户实时交流工作进展。
主要特征
新的工作流
- 在开始页面 新添一组“新陈代谢″工作流模块
- 流平衡分析用以识别酶,反应和代谢产物,包括聚类和流数据映射
- 丰富的上游分析(TRANSFAC®和TRANSPATH®)
- 集中的上游分析(TRANSFAC®和TRANSPATH®)
- 带有反馈回路的上游分析(TRANSFAC®和TRANSPATH®)表明主调节剂具有来自输入组的表达值(倍数变化)
- 识别组织特定轨迹中的丰富基序(TRANSFAC®)
- 跨物种鉴定启动子中的丰富基序
- 跨物种映射到本体
更新的数据库
- 人类代谢反应(HMR):HMR2包含3,765个基因,与8,000多个反应和3,000多个独特代谢产物相关。
- 人类新陈代谢的整体重建(Recon 2):最全面的代表人类新陈代谢已可用计算机建模
- 集成最新版本的TRANSFAC®和TRANSPATH®(3版)
- HOmo sapiens COmprehensive MOdel COllection(HOCOMOCO):此数据库部分基于TRANSFAC,提供了601个人类TF和396个小鼠TF的转录因子(TF)结合模型。
- Reactome数据库已更新至版本63。
- 集成了新的Ensembl版本(发行版96),Ensembl Homo sapiens(hg38),Ensembl Mus musculus(mm10),Ensembl Rattus norvegicus(rn6)
新方法

Subread – 对齐DNA和RNA序列读取

Subjunc – 外显子–外显子交界

featureCounts – 读取计数

exactSNP – 个体样品的单核苷酸多态性

Limma voom – 差异表达分析

Guided limma – 线性模型分析

HISAT2 – 快速灵敏的对准工具

HTseq Count – 读取每个功能的映射

HTseq QA – 质量报告

EdgeR – R中数字化基因表达数据的经验分析

Plotting – 饼图和条形图

Tree map – 减少功能分类术语

获取miRNA靶标

miRNA前馈环

分析miRNA富集靶标
相关资料下载
- 6.2 版本的特征列表 下载PDF
- 用户指南 5.0版 在线版
- 用户指南 5.0版 下载PDF
- 用户指南 5.0版 下载EPUB
- 5.0 版本的特征列表 下载PDF
- GeneXplain平台宣传册 下载PDF
- 快速入门指南 下载PDF
![]()
产品咨询
想获得 geneXplain 平台的免费试用或询问产品报价并购买geneXplain platform许可证,请点击 此处
![]()
近期出版物
Mekonnen, Y. A., Gültas, M., Effa, K., Hanotte, O. and Schmitt, A. O. (2019) Identification of Candidate Signature Genes and Key Regulators Associated With Trypanotolerance in the Sheko Breed. Front. Genet. 10:1095. doi: 10.3389/fgene.2019.01095. Link
Blazquez R., Wlochowitz D., Wolff A., Seitz S., Wachter A., Perera-Bel J., Bleckmann A., Beißbarth T., Salinas G., Riemenschneider M.J., Proescholdt M., Evert M., Utpatel K., Siam L., Schatlo B., Balkenhol M., Stadelmann C., Schildhaus H.U., Korf U., Reinz E., Wiemann S., Vollmer E., Schulz M., Ritter U., Hanisch UK., Pukrop T. (2018) PI3K: A master regulator of brain metastasis-promoting macrophages/microglia. Glia. 66(11):2438-2455. doi: 10.1002/glia.23485. Epub 2018 Oct 25. Link
Orekhov, A. N., Oishi, Y., Nikiforov, N. G., Zhelankin, A. V., Dubrovsky, L, Sobenin, I. A., Kel, A., Stelmashenko, D., Makeev, V. J., Foxx, K., Jin, X., Kruth, H. S. and Bukrinsky, M. (2018) Modified Ldl Particles Activate Inflammatory Pathways In Monocyte-Derived Macrophages: Transcriptome Analysis. Curr. Pharm. Des., 11. doi: 10.2174/1381612824666180911120039. Link
Kalozoumi, G., Kel-Margoulis, O., Vafiadaki, E., Greenberg, D., Bernard, H., Soreq, H., Depaulis, A., Sanoudou, D. (2018) Glial responses during epileptogenesis in Mus musculus point to potential therapeutic targets. PLoS One. 13(8):e0201742. doi: 10.1371/journal.pone.0201742. Link
Smetanina, M.A., Kel, A.E., Sevost’ianova, K.S., Maiborodin, I.V., Shevela, A.I., Zolotukhin, I.A., Stegmaier, P., Filipenko, M.L. (2018) DNA methylation and gene expression profiling reveal MFAP5 as a regulatory driver of extracellular matrix remodeling in varicose vein disease. Epigenomics. 10(8):1103-1119. doi: 10.2217/epi-2018-0001. Link
Boyarskikh, U., Pintus, S., Mandrik, N., Stelmashenko, D., Kiselev, I., Evshin, I., Sharipov, R., Stegmaier, P., Kolpakov, F., Filipenko, M., Kel, A. (2018) Computational master-regulator search reveals mTOR and PI3K pathways responsible for low sensitivity of NCI-H292 and A427 lung cancer cell lines to cytotoxic action of p53 activator Nutlin-3. BMC Med Genomics. 11(Suppl 1):12. doi: 10.1186/s12920-018-0330-5. Link
Triska, M., Solovyev, V., Baranova, A., Kel, A., Tatarinova, T.V. (2017) Nucleotide patterns aiding in prediction of eukaryotic promoters. PLoS One. 12(11):e0187243. doi: 10.1371/journal.pone.0187243. Link
Niehof, M., Hildebrandt, T., Danov, O., Arndt, K., Koschmann, J., Dahlmann, F., Hansen, T. and Sewald, K. (2017) RNA isolation from precision-cut lung slices (PCLS) from different species. BMC Res. Notes 10, 121. doi: 10.1186/s13104-017-2447-6. Link
Mandić, A. D., Bennek, E., Verdier, J., Zhang, K., Roubrocks, S., Davis, R. J., Denecke, B., Gassler, N., Streetz, K., Kel, A., Hornef, M., Cubero, F. J., Trautwein, C. and Sellge, G. (2017) c-Jun N-terminal kinase 2 promotes enterocyte survival and goblet cell differentiation in the inflamed intestine. Mucosal Immunol. 10(5):1211-1223. doi: 10.1038/mi.2016.125. Link
Pietrzyńska, M., Zembrzuska, J., Tomczak, R., Mikołajczyk, J., Rusińska-Roszak, D., Voelkel, A., Buchwald, T., Jampílek, J., Lukáč, M., Devínsky, F. (2016) Experimental and in silico investigations of organic phosphates and phosphonates sorption on polymer-ceramic monolithic materials and hydroxyapatite. Eur. J. Pharm. Sci. 93, 295-303. doi: 10.1016/j.ejps.2016.08.033. Link
Kural, K. C., Tandon, N., Skoblov, M., Kel-Margoulis, O. V. and Baranova, A. V. (2016) Pathways of aging: comparative analysis of gene signatures in replicative senescence and stress induced premature senescence. BMC Genomics 17(Suppl 14), 1030. doi: 10.1186/s12864-016-3352-4. Link
Kel, A. E., Stegmaier, P., Valeev, T., Koschmann, J., Poroikov, V., Kel-Margoulis, O. V. and Wingender, E. (2016) Multi-omics “upstream analysis” of regulatory genomic regions helps identifying targets against methotrexate resistance of colon cancer. EuPA Open Proteomics 13, 1-13. doi: 10.1016/j.euprot.2016.09.002. Link
Ciribilli, Y., Singh, P., Inga, A., Borlak, J. (2016) c-Myc targeted regulators of cell metabolism in a transgenic mouse model of papillary lung adenocarcinoma. Oncotarget 7, 65514-65539. doi: 10.18632/oncotarget.11804. Link
Wlochowitz, D., Haubrock, M., Arackal, J., Bleckmann, A., Wolff, A., Beißbarth, T., Wingender, E., Gültas, M. (2016) Computational Identification of Key Regulators in Two Different Colorectal Cancer Cell Lines. Front. Genet. 7, 42. doi: 10.3389/fgene.2016.00042. Link
Lee, E.H., Oh, J.H., Selvaraj, S., Park, S.M., Choi, M.S., Spanel, R., Yoon, S. and Borlak, J. (2016) Immunogenomics reveal molecular circuits of diclofenac induced liver injury in mice. Oncotarget 7, 14983-15017. doi:10.18632/oncotarget.7698. Link
Borlak, J., Singh, P. and Gazzana, G. (2015) Proteome mapping of epidermal growth factor induced hepatocellular carcinomas identifies novel cell metabolism targets and mitogen activated protein kinase signalling events. BMC Genomics 16, 124. doi:10.1186/s12864-015-1312-z. Link
Koschmann, J., Bhar, A., Stegmaier,P., Kel, A. E. and Wingender, E. (2015) “Upstream Analysis”: An integrated promoter-pathway analysis approach to causal interpretation of microarray data. Microarrays 4, 270-286. doi:10.3390/microarrays4020270. Link
Shi, Y., Nikulenkov, F., Zawacka-Pankau, J., Li, H., Gabdoulline, R., Xu, J., Eriksson, S., Hedström, E., Issaeva, N., Kel, A., Arnér, E.S., Selivanova, G. (2014) ROS-dependent activation of JNK converts p53 into an efficient inhibitor of oncogenes leading to robust apoptosis. Cell Death Differ. 21, 612-623. doi:10.1038/cdd.2013.186 Link
Schlereth, K., Heyl, C., Krampitz, A.M., Mernberger, M., Finkernagel, F., Scharfe, M., Jarek, M., Leich, E., Rosenwald, A., Stiewe, T. (2013) Characterization of the p53 Cistrome – DNA Binding Cooperativity Dissects p53’s Tumor Suppressor Functions. PLoS Genet. 9, e1003726. PubMed
Nikulenkov, F., Spinnler, C., Li, H., Tonelli, C., Shi, Y., Turunen, M., Kivioja, T., Ignatiev, I., Kel, A., Taipale, J., Selivanova, G. (2012) Insights into p53 transcriptional function via genome-wide chromatin occupancy and gene expression analysis. Cell Death Differ. 19, 1992-2002. PubMed
Zawacka-Pankau, J., Grinkevich, V.V., Hunten, S., Nikulenkov, F., Gluch, A., Li, H., Enge, M., Kel, A., Selivanova, G. (2011) Inhibition of glycolytic enzymes mediated by pharmacologically activated p53: targeting Warburg effect to fight cancer. J. Biol. Chem. 286, 41600-41615. PubMed
Kel, A., Kolpakov, F., Poroikov, V., Selivanova, G. (2011) GeneXplain — Identification of Causal Biomarkers and Drug Targets in Personalized Cancer Pathways. J. Biomol. Tech. 22(suppl.), S16. PubMed


